La professeure émérite de l'Institut du cancer Rosalind et Morris Goodman (ICG), Nicole Beauchemin, Ph.D., a joué un rôle essentiel dans les recherches révolutionnaires sur le cancer colorectal menées à l'ICG. Lisez son article ci-dessous pour en savoir plus sur l'héritage de l'ICG dans l'avancement de la recherche sur le cancer colorectal.
Le cancer colorectal (CCR) est un domaine de recherche de longue date à l'Institut du cancer Goodman depuis que le Dr Phil Gold, fondateur du Centre du cancer de McGill, a entamé ses projets de recherche visant à identifier des antigènes tumoraux spécifiques servant de biomarqueurs, permettant de suivre l'évolution de la maladie ou de cibles pour des thérapies spécifiques. Cela a conduit à l'identification de l'antigène carcinoembryonnaire (CEA) et à l'utilisation ultérieure d'anticorps monoclonaux spécifiques comme outils de détection en milieu clinique (1). La surexpression de l'CEA dans les tumeurs par rapport aux tissus normaux avec des anticorps anti-CEA spécifiques a démontré une très haute sensibilité (2). Les taux préopératoires du CEA sérique étaient élevés lorsqu'ils ont été testés dans une cohorte de plus de 17 000 patients; ce test a confirmé un risque accru de 60 % de mortalité globale à tous les stades du cancer (3). Le CEA est donc devenu un excellent biomarqueur utilisé cliniquement dans le monde entier pour surveiller la progression du cancer colorectal. De plus, la caractérisation de l'ADNc de CEA en 1987 par plusieurs équipes internationales, dont le groupe de recherche du Pr. Stanners, a abouti à une découverte inattendue: le CEA était le signe distinctif d'une grande famille de gènes (la famille de gènes CEACAM) comptant jusqu'à 22 membres, tous porteurs de domaines caractéristiques semblables à ceux des immunoglobulines (4).
Les trois membres les plus importants de la famille CEACAM impliqués dans le cancer colorectal sont le CEA, le CEACAM6 et le CEACAM1 (figure 1). Ils fonctionnent tous comme des molécules d'adhésion intercellulaire (4). Outre le CEA, d'autres études ont confirmé que CEACAM6 joue également un rôle dans la progression du cancer colorectal, son expression étant régulée à la hausse depuis les adénomes jusqu'aux tumeurs malignes ; son expression élevée est statistiquement associée à une survie globale et à une survie sans maladie plus faibles (5). Les chercheurs Chan et Stanners (ICG) ont développé un modèle de souris transgénique CEABAC, contenant un chromosome artificiel bactérien portant les gènes humains CEA, CEACAM6 et CEACAM7, qui surexprime les protéines CEA et CEACAM6 de 2 à 20 fois dans le côlon. Cela a conduit à une désorganisation importante du tissu colorectal (hyperplasie et dysplasie) et lorsque le CCR a été provoqué avec de l’agent cancérigène azoxyméthane, les tumeurs du côlon étaient plus importantes en nombre et en taille par rapport à celles apparaissant chez les souris contrôle (Figure 1) (6).
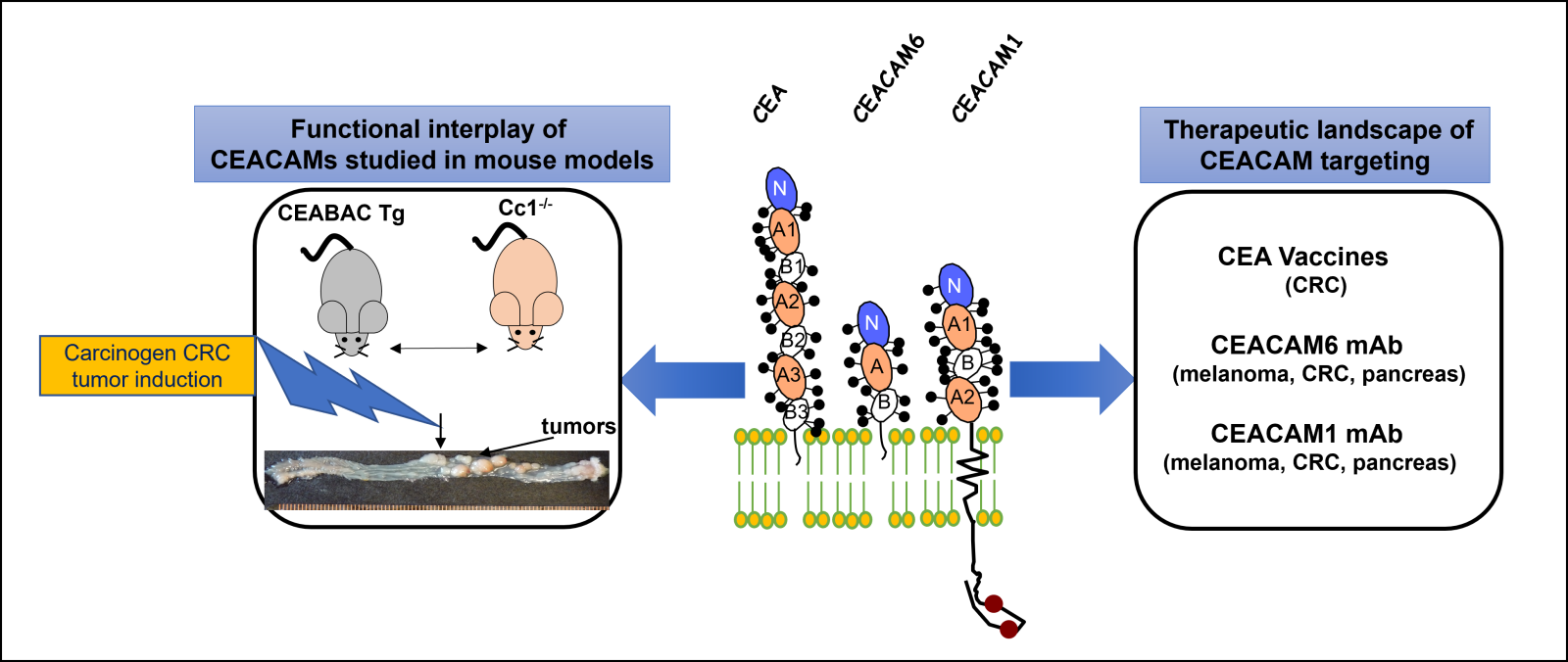
Figure 1 : Le cancer colorectal (CRC) a été largement étudié à l’Institut du Cancer Rosalind et Morris Goodman au fil des années. Ces études ont conduit à l’identification de l’Antigène Carcinoembryonnaire (CEA), un biomarqueur largement utilisé pour le CRC, mais aussi à la découverte d’une grande famille de gènes codant pour les protéines CEACAM (CEA Cell Adhesion Molecules), parmi lesquelles CEACAM6 et CEACAM1 jouent également un rôle clé dans le développement et la progression du CRC (PANNEAU CENTRAL). Les protéines CEACAM possèdent des domaines immunoglobulines et sont hautement glycosylées (bâtons), avec soit un lien phosphatidyl-inositol (CEA et CEACAM6), soit un domaine transmembranaire et un long domaine cytoplasmique (CEACAM1) contenant deux résidus Tyr-phosphorylables (boules rouges). Pour mieux comprendre le rôle de CEA, CEACAM6 et CEACAM1 dans la progression du CRC, plusieurs modèles murins ont été développés, incluant une surexpression des gènes CEA, CEACAM6 et CEACAM7 (souris CEABAC) ou une déficience en CEACAM1 (souris Cci1-/-). Lorsqu’ils sont traités avec un agent carcinogène spécifique au CRC, des tumeurs CRC se développent et sont analysées (PANNEAU GAUCHE). Au fil des années, de nombreuses approches thérapeutiques ont été explorées par les groupes de recherche internationaux sur les CEACAM, et certaines sont encore en cours de validation clinique (PANNEAU DROIT).
Notre propre groupe a concentré ses recherches sur la protéine CEACAM1, un autre membre de la même famille, mais avec des propriétés très différentes et des profils d'expression plus larges. Elle est présente sur de nombreux types de tumeurs, y compris le CCR, mais aussi à la surface des petits vaisseaux sanguins, des ganglions lymphatiques et de nombreuses cellules immunitaires (4). CEACAM1 est également une protéine d'adhésion cellulaire, mais elle projette dans le cytoplasme l'un de ses deux domaines cytoplasmiques, dont le plus long (CEACAM1-L) s'avère être tyrosine-phosphorylé par une variété de Tyr kinases; cela conduit à des interactions de haute affinité avec les Tyr phosphatases SHP-1 et SHP-2 et à l'activation de nombreuses cascades de signalisation (4). Dans le développement du CCR chez l'homme, l'expression de CEACAM1 a été initialement signalée comme étant régulée à la baisse (contrairement à la régulation à la hausse de CEA et CEACAM6) dans les adénomes, la phase précoce du CCR. Pour confirmer que sa régulation était significative dans le développement du CCR, nous avons développé un modèle de souris Ceacam1-/-; la suppression de CEACAM1 a entraîné une prolifération accrue des cellules du côlon par rapport aux contrôles, les cellules coliques présentant une diminution de la mort cellulaire programmée. Lorsque le cancer du côlon est déclenché par un traitement cancérigène à l'azoxyméthane, les souris Ceacam1-/- produisent un grand nombre de tumeurs du côlon (7). Dans un second modèle de CCR, le modèle de souris Ceacam1-/- X Apc1638N/+, l'élimination de l'expression de CEACAM1 a augmenté la taille et le nombre des tumeurs du CCR (8). Cependant, des données cliniques récentes indiquent que l'expression de CEACAM1 augmente avec le niveau de dysplasie du côlon (9); il reste possible que l'équilibre entre les domaines cytoplasmiques courts et longs de CEACAM1 joue un rôle important dans cette divergence (9). Paradoxalement, les stades avancés du CCR présentent une forte expression de CEACAM1 qui est associée au caractère invasif et aux métastases du foie (10-13).
Avec l'arrivée de l'immunothérapie en tant que thérapie anticancéreuse efficace, CEACAM1 a été reconnu comme une cible clinique anticancéreuse en raison de sa forte expression dans les cellules immunitaires. Il est présent à la surface des lymphocytes T CD8+ circulants et des lymphocytes infiltrant les tumeurs du CCR, ainsi que d'autres cellules immunitaires (14). La disponibilité d'un anticorps monoclonal hautement spécifique reconnaissant uniquement le membre CEACAM1 (à l'exclusion de tous les autres CEACAM) a montré que le blocage de l'activité du CEACAM1 à l'aide de cet anticorps arrête le développement du cancer colorectal et d'autres cancers et les fait même régresser. L'inhibition de l'activité de CEACAM1 le désengage du contact physique avec deux autres régulateurs importants des cellules immunitaires, principalement TIM-3 et PD-1, ce qui mène à la libération des cellules T du frein imposé par TIM-3 ou PD-1 et réactive ces cellules T pour combattre de nombreuses formes de cancer, y compris le CCR (14, 15). De futurs essais cliniques pourraient valider son utilité pour combattre la progression du cancer chez les patients humains. La traduction clinique dans ce domaine a été ralentie par le manque de connaissances sur la façon dont ces trois membres de la famille CEACAM interagissent entre eux à la surface des cellules normales et malignes du CCR, mais aussi entre ceux exprimés sur les cellules tumorales et les cellules immunitaires infiltrant la tumeur (16, 17).
Le groupe de Philippe Gros a également fait des progrès significatifs dans la compréhension d'une autre forme de la maladie, le CCR associé à la colite (CA-CRC), en identifiant des loci de gènes à haut risque dans des modèles de souris (Ccs3 et Ccs5) et le gène du facteur de régulation de l'interféron 1 (Irf1) (18,19). La suppression du gène Irf1 provoque une inflammation importante et l'infiltration de cellules immunitaires myéloïdes, granulocytaires et lymphoïdes dans le côlon des souris, avec un développement tumoral important lorsqu'il est déclenché par des traitements cancérigènes. Les mêmes changements transcriptionnels associés à la délétion d'Irf1 chez la souris sont retrouvés dans des échantillons cliniques humains de cancers CCR de stade 3 et 4 et associés à un pronostic plus défavorable (19). Ces résultats mettent en évidence un grand nombre de biomarqueurs possibles utilisés pour évaluer la progression du CCR chez les patients et patientes, afin d'offrir à ces derniers des traitements agressifs appropriés en temps opportun.
Article rédigé par Nicole Beauchemin, Ph.D.
Références: (les noms en gras correspondent aux membres de l’Institut du cancer Rosalind et Morris Goodman)
1. Gold P, Freedman SO. (1965); PMID: 4953873
2. Tiernan, J.P. et al., (2013); PMID; 23322207
3. Thirunavukarasu, P. et al. (2011); PMID: 21421861
4. Beauchemin, N., and Arabzadeh, A. (2013); PMID: 23903773
5. Guanhua Wu, et al. (2024); PMID: 38240103
6. Chan CH, Cook D, Stanners CP. (2006); PMID: 16632476
7. Leung, N….Beauchemin N. (2006); PMID: 16619040
8. Leung, N….Beauchemin N. (2008); PMID: 18454175
9. Götz, L et al. (2024); PMID: 39674877
10. Ieda, J. et al. (2011); PMID: 21413011
11. Yeatman, T.J. et al. (1997); PMID:9834361
12. Kang, W.Y. et al. (2007); PMID:17143599
13. Song, J. H. et al. (2011); PMID:20524097
14. Huang, Y.H……Beauchemin, N., Blumberg, R.S. (2024); PMID: 38956268
15. Huang, Y.H ….Beauchemin, N. …Blumberg, R.S. (2015); PMID: 25363763
16. Stern, N. et al. (2005); PMID: 15905509
17. Pinkert, J. et al. (2022); PMID: 35141051
18. Meunier, C……Beauchemin, N., Gros, P. (2010); PMID: 19915610
19. Jeyakumar, T. ….Beauchemin, N., Gros, P. (2019); PMID: 31827213